
时间:2022-04-27作者:

| 摘要 |
基于结构的药物发现(Structure-based drug discovery, SBDD)是设计和优化创新药的必要方法。本篇综述将深入探讨冷冻电镜(cryo-EM)在SBDD领域中的快速崛起及它的主要作用,以及阐释它如何为高价值药理学靶点提供丰富的全新结构信息。冷冻电镜技术相比X射线晶体学的主要优势在于,它可以跳过繁琐的结晶步骤,从而直接对玻璃化的生物大分子进行成像;冷冻电镜也可以提供更多维度的信息,包括异质性和动态性。此外,本综述还将讨论冷冻电镜近期和未来的发展,并探讨该技术将在SBDD的管线中产生何种广泛的影响。
冷冻电镜时代的SBDD
SBDD是一种基于靶点的原子级结构基础信息,针对该靶点进行理性药物设计的研发方法。20世纪80年代,随着Captopril卡托普利和多佐胺Dorzolamide等酶靶向药物[1-3]获批上市,SBDD方法初露锋芒。这一批由FDA批准的药物结合了晶体结构模型与计算机辅助分子建模这两大新兴技术,并成功解决了传统湿实验室的高通量筛选方法(HTS)所面临的昂贵、耗时及低回报率等问题。此后,随着计算技术的不断革新,大量药物靶点的晶体结构得以解析,SBDD方法进入了飞速发展阶段。从1999年到2013年,在113个获批的first-in-class药物中,有78个是基于SBDD方法发现的[4]。尽管SBDD的发展足够迅速,但学界及制药行业内对它的期望显然更高。SBDD方法往往能另辟蹊径,对过往认为不可成药的靶点进行验证,并进一步开发新药。如K-Ras(G12C)靶点,它利用晶体学结构确定了一个以前未知的结合口袋,以避免与皮摩尔亲和力的GDP/GTP竞争[5]。由于靶点验证是发现和开发工作中的主要难题之一,first-in-class药物分子可以为靶点的有效性和疾病应用提供新的见解,例如bromodomain溴结构域抑制剂(+)-JQ-1和I-BET762,这些化合物被成功用于表征和验证溴结构域在各种疾病中的重要性,并催生了大量的临床候选药物[6]。即使是FDA批准的已知药物靶点,临床上也常常需要进一步的SBDD,比如有些药物需要进行更好的选择性的优化设计。厄菲替尼(erdafitinib)在经过针对性的设计改造后,表现出了相对于原先药物对成纤维生长因子受体更高的选择[7]。此外,有一些药物可能需要优化效力或疗效,或提供特定受体亚型的选择性,如改善鞘氨醇-1-磷酸(S1P)抑制剂西波尼莫德(siponimod)对S1P1而非对S1P3的选择性,是提高其在疗效和安全性上优于非选择性S1P抑制剂的关键[8]。该药物靶向S1P1,而非S1P3,此外,许多抗病毒、抗菌和抗癌药物正面临着抗药性问题,SBDD方法能够基于产生耐药性的靶点结构,对药物进行持续改进[9-11]。SBDD工作的瓶颈在于获取高分辨率的生物靶点结构信息。虽然一些小而有序的生物分子满足X射线晶体学的研究范畴,但大部分已知靶点中的蛋白质,例如跨膜受体或动态复合物,都难以结晶,导致这些靶点蛋白无法利用晶体手段进行高分辨率结构解析。此外,X射线晶体学往往会对靶点蛋白进行改造,如进行截短体设计、引入热稳定性突变或插入一段外源的结构域,从而影响后续的SBDD结构信息分析。还需要考虑的一个关键因素是,大量的靶点蛋白性质上达不到结晶的条件要求。不过,上述的这些难点正被冷冻电镜技术逐一攻克。冷冻电镜技术的分辨率已足够高,其产生的大量数据也可用于计算辅助药物设计(CADD)方法,这也是本综述的核心议题。与X射线晶体学不同的是,冷冻电镜无需对目标靶点进行结晶:纯化过的靶点生物大分子会被瞬间冻结在一层薄薄的非结晶玻璃体冰中,再经由透射电镜成像以记录下几十万到几百万个冷冻电镜颗粒数据,用于重构三维静电势图并对大分子进行精确建模(见[12]关于冷冻电镜的回顾)。因此,这种技术很适合于蛋白质复合物、热稳定性较低和动态运动较高的蛋白质以及脂质胶束中的跨膜蛋白质的结构测定。随着分辨率的不断提高,冷冻电镜已经成为药物设计的强大工具。
冷冻电镜与药物发现
在2014年之前,冷冻电镜几乎无法解析出优于4.0Å分辨率的结构,这直接导致它无法对SBDD工作提供有效的数据支持。然而,在过去的几年里,冷冻电镜方法的爆炸性突破产出了大量高分辨率的结构数据,这在以前是无法实现的。这一质的飞跃要归功于许多技术革新,如用于记录图像的直接电子探测器、改进的计算方法和处理大型数据集的硬件集群,这些技术的飞跃在其他文献内有详细回顾[13]。此外,作为一种直接可视化的技术,冷冻电镜能够快速判断样品的聚集性和稳定性等问题,从而通过遗传和生物化学手段,用互作因子稳定蛋白、或通过优化去垢剂从细胞膜环境中提取膜蛋白等方法来快速改善样品质量。综合以上,在PDB中的分辨率为4.0Å或更高的冷冻电镜结构的数量已经从2014年之前的合计16个增长到仅2020年一年提交1753个新结构的规模(图1, A)。在新上传的结构中,分辨率高于4.0和3.5 Å的比例分别从2015年的36%和12%增加到2020年的75%和50%。更振奋人心的是,截止2020年,分辨率高于3.0Å和2.5Å的冷冻电镜结构比例,分别达到了18%和3%,实现了冷冻电镜结构解析前所未来的突破(图1, B)。为了系统评估冷冻电镜对SBDD领域的影响,我们(作者)调查了2018年美国200种最常用处方药的靶点相关结构数据[14]。72%的靶点在PDB数据库中含有结构信息。细分而言,这些结构信息是通过X射线晶体学技术(42%)、冷冻电镜技术(15%)或两者结合(15%)而确定的(图1, C)。通过冷冻电镜技术解析的靶点涵盖了许多跨膜蛋白,如离子通道(GABAA、CaV、NaV和KATP)、激活态的G蛋白偶联受体(GPCRs)和转运体蛋白(5-羟色胺转运体、NaCl转运体)。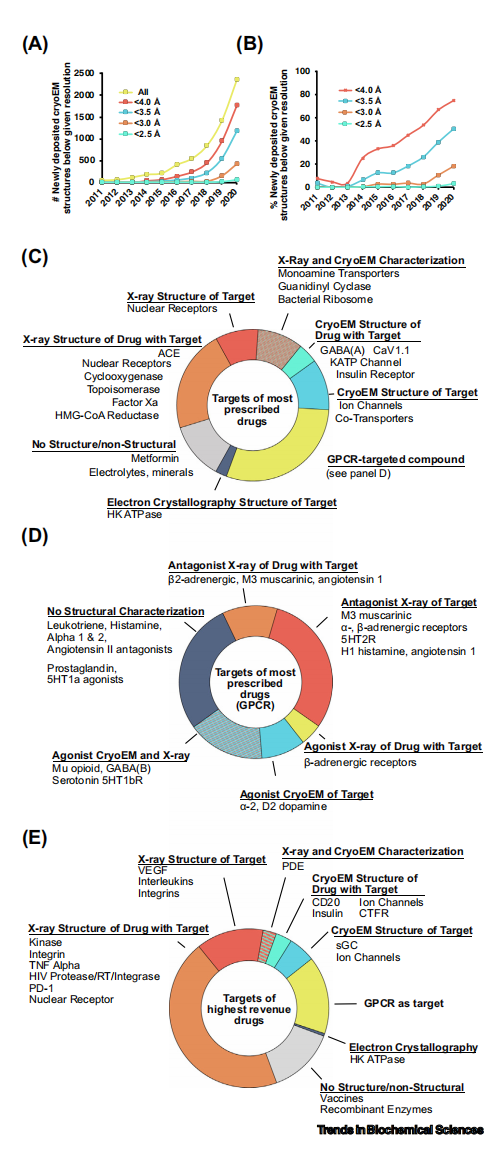
图1.冷冻电镜分辨率的提高及其对蛋白质药物结构表征的贡献。(A) PDB中上传的低于特定分辨率的冷冻电镜结构的绝对数量;(B) PDB中上传的低于特定分辨率的冷冻电镜结构的百分比的[61]。(C)2018年200个热门处方药的靶点图,按靶点的结构特征分类;(D)44个热门GPCRs处方药的靶点图,按结构特征分类;(E)2018年200个销量最高的药物的靶点图(作为新药的代表),按靶点的结构特征分类。2020年的数据是由Njardarson实验室公示的2018年200种最受欢迎的处方药和200种销量最高的药物的蛋白质靶点(如果适用),然后在PDB中确定相关结构,进行人工筛选。
在200多种最常见的处方药中,GPCRs占据了44种,这些药物包括靶向GPCRs的激动剂、拮抗剂和反向激动剂(图1, D;注意,拮抗剂和反激动剂在药理学上不同,但在这里我们(作者)把它们统一归为拮抗剂)。这些GPCRs中的32个(73%)已经进行了某种形式的结构解析,包括与拮抗剂(44%)或激动剂(7%)结合的晶体结构,与激动剂(9%)结合的冷冻电镜结构,或由X射线晶体学和冷冻电镜手段共同进行的结构解析(20%)。值得注意的是,GPCR的高度动态结构使其难以获得高质量的晶体,因此大多数的GPCR晶体结构都是与拮抗剂结合后才得以进行结构解析的。综上所述,冷冻电镜技术在针对市场上已经存在多年的处方药中中具有深刻影响。为了更加深入了解冷冻电镜技术在未来药物发现中的作用,我们(作者)还调查了2018年取得最高利润的200种药物,以代表那些市面上新进发现的药物[14](图1, E),我们简称新药。这批新药和之前提到的那些最常用的药物之间存在明显的差异。相当一部分新药已经用晶体学进行了表征,反映了结构数据在当今药物研发工作中的重要性:即便不是由结构驱动的,也很少有不追求结构的情况,因为结构信息可以为先导化合物的优化和进一步发现提供关键数据。此外,考虑到漫长的药物开发时间,冷冻电镜这一最近几年才崛起的新技术在这份名单中的占比虽小,但贡献仍相当可观。这些药物和靶点包括生物制药、离子通道和GPCRs,以及其他不适合结晶的高活性大分子。
冷冻电镜对SBDD的贡献解析新型结构
虽然有许多FDA批准的药物靶点结构可被X射线晶体学解析,冷冻电镜正在为越来越多的难结晶、甚至不可结晶的靶点打开大门,如分子量更大、更动态的蛋白质和蛋白质复合物。冷冻电镜也显著降低了对细胞内复合体的研究难度,如病原体的核糖体、染色质修饰复合体和转录机器[15-17]。例如冷冻电镜技术近期解析了一种与线粒体体RNA聚合酶复合体相关的first-in-class 抑制剂的结构[18]。值得注意的是,在膜蛋白领域,冷冻电镜的贡献无可比拟。不管是传统的药物,还是新型处方药,很多药物靶向针对GPCRs、离子通道和转运体蛋白。然而,利用X射线晶体学手段来解析膜蛋白的结构非常困难。尽管脂质立方结晶在GPCR领域取得了一些进展,但在结晶过程中,GPCR蛋白通常需要进行热稳定突变,或融合其他蛋白进行改造,以促进晶体的形成。并且,为了获取某种改造后的稳定的构象,还需要对克隆构建、实验方法及条件进行大量繁琐复杂的筛选[19, 20]。相比之下,冷冻电镜结构可以直接用来解析经过去污剂或纳米盘处理后的在生化上性质稳定的膜蛋白,并获得处于或者接近生理状态的蛋白的结构。冷冻电镜的在解析庞杂的膜蛋白的结构中能力势不可挡,并且已有大量的高分辨率结构被成功解析。长久以来膜蛋白一直都是获批药物的热门靶点,它们的结构也只是近期才被冷冻电镜揭示(图2)。
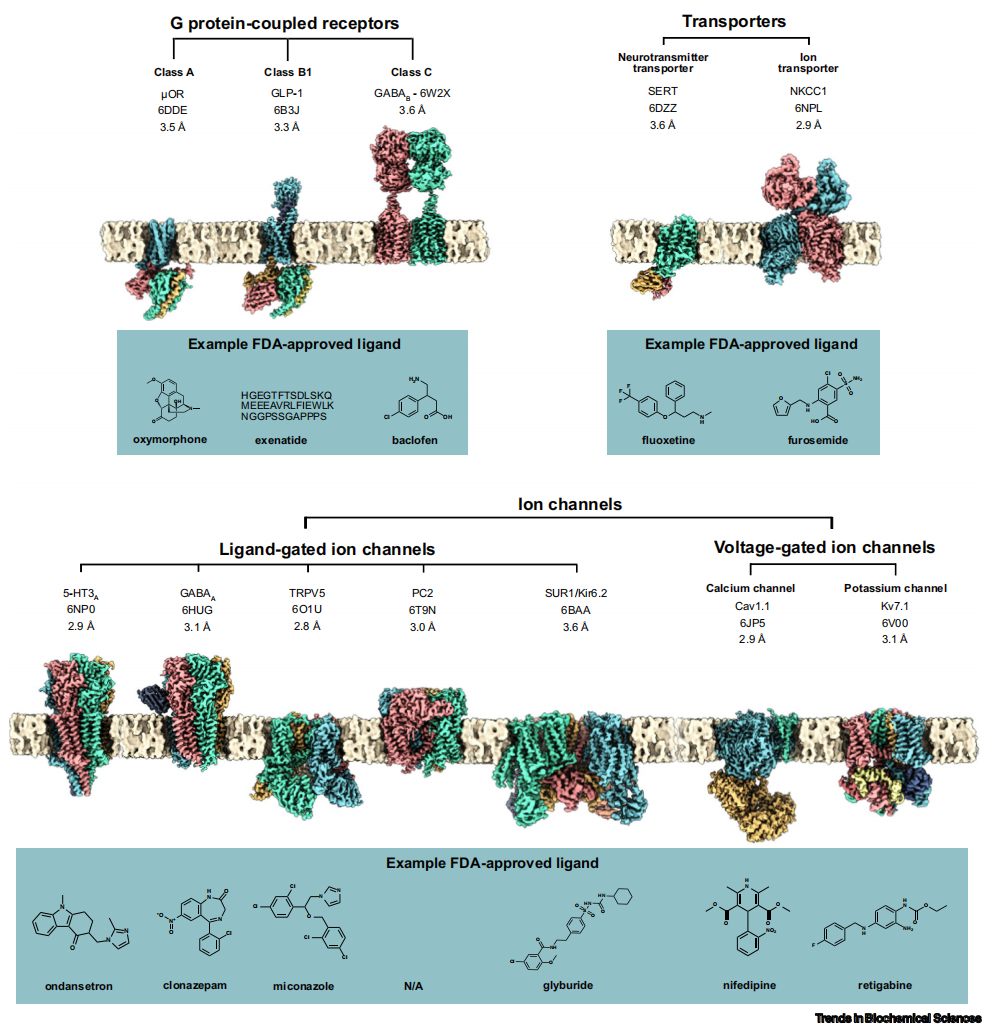
图2. G蛋白偶联受体、转运体(上排)和离子通道(下排),每个受体有相应的FDA批准的配体分子(蓝框)。
利用冷冻电镜解析膜蛋白结构的突出进展,部分原因受益于新试剂的设计和使用。这些试剂可以在体外纯化过程中维持跨膜蛋白的结构,在冷冻制样过程中保护蛋白,并为高分辨率的结构解析提供均质样品。去垢剂如正十二烷基β-D-麦芽糖苷(DDM)和月桂基麦芽糖新戊二醇(LMNG),可以有效地从细胞膜上溶解跨膜蛋白,并维持蛋白质的生理状态构象。去垢剂的使用也会产生一些问题,如去垢剂形成的空胶束和与包裹蛋白质的去垢剂同时存在存在会引起样品的不均一,对后期的数据处理处理产生影响;也可能会导致冷冻样品制备时的气液界面收到破坏,产生一些不好的结果[21]。脂质纳米盘是去垢剂的一种替代品,原则上可以为结构和生物物理研究提供接近胜利状态的脂质双分子层。脂质纳米盘在膜蛋白药物靶点上的应用已经非常关键和广泛。举例而言,将纳米盘与冷冻电镜技术相结合,成功阐明了TRPV1和TRPV5离子通道(在TRPV1的情况下,脂质对抑制剂的结合至关重要)、GABAA配体门控离子通道、人类P-糖蛋白以及GPCR-β-arrestin复合物的高分辨率结构和机制[22-26]。关于纳米盘的进一步介绍可查阅[27, 28]。冷冻电镜还可以用来解析嵌入脂质体中的蛋白质的结构,允许在更接近生理状态的的电化学梯度中对离子通道以及孔蛋白进行可视化研究[29, 30]。在过去的几年中,冷冻电镜也在生物制药领域产生了巨大影响。在较新的药物中,生物制药的占比正越来越高。如果仅将目光聚焦于药物靶点识别这一领域,生物制药的结晶技术确实称得上有所改善。然而,冷冻电镜已经为一些关键的生药物研发提供了基于全长蛋白的结构信细节息胰岛素受体一种二聚化的酪氨酸激酶受体蛋白,在调节人体的葡萄糖平衡方面起着关键作用。胰岛素受体信号通路的失调会引起一些疾病,如II型糖尿病,全球约有9.3%(4.63亿人)受到影响[31]两个独立的研究小组利用冷冻电镜在胰岛素受体结构解析方面取得了突破进展;第一个小组以4.3Å和7.2Å的分辨率分别解析了与一个或两个胰岛素分子结合的胰岛素受体胞外结构域结构,第二个小组以3.1Å的分辨率获得了与四个胰岛素分子结合的胰岛素受体胞外结构域结构(图3, A)。这些结构解释了胰岛素受体结合胰岛素的不同结合位点,以及激活这一关键药物靶点所进行的构象变化[32, 33]。
类似的例子比比皆是:从HER2-trastuzamab-pertuzumab复合物到SARS-CoV-2和中和抗体的结构解析,冷冻电镜为生物治疗的新老靶点提供了新的视点,为进一步发现和开发仿制药和first-in-class药物铺平了道路。另一个值得注意的例子是B淋巴细胞抗原CD20,它是治疗白血病和自身免疫性疾病的一个重要的治疗靶点,尽管其功能作用仍不清楚。尽管CD20的分子量较小,只要35kDa左右,但分别与单克隆抗体利妥昔单抗(rituximab)、奥法图单抗(ofatumumab)和奥比努单抗(obinutuzumab)的Fab结合形成复合物后,都解析获得分辨率较高的CD20复合物结构(图3, B)[36, 37]。负染结果显示,利妥昔单抗与CD20结合后,可诱导形成高度有序的高级结构[36],这一发现对激活先天免疫的补体系统提供了全新见解。由于复合物中的高度动态和跨膜结构域的存在,利用结晶手段结构解析几乎不可能实现,冷冻电镜技术的应用实现了这一可能。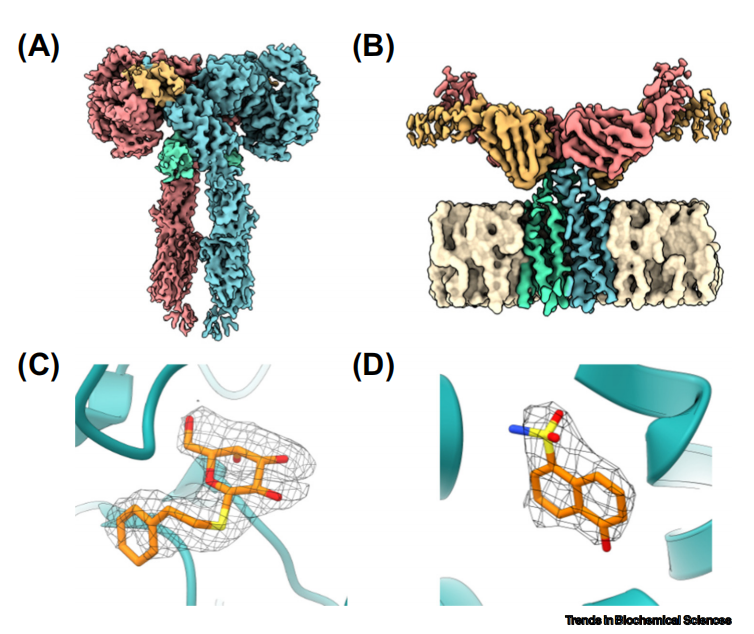
图3.冷冻电镜(cryo-EM)在小分子和生物制药发现方面的效用。(A)与胰岛素结合的胰岛素受体(PDB ID 6PXV)和(B)CD20与利妥昔单抗复合物(PDB ID 6VJA)冷冻电镜密度图。(C)使用GemSpot(PDB ID 6CVM)将小分子PETG精确地建模到β-半乳糖苷酶的冷冻电镜图像中。(D)基于片段的PKM2的发现,冷冻电镜密度允许正确识别和放置发现片段(PDB ID:6TTF)
尽管冷冻电镜在膜蛋白结构测定领域已经迈出了一大步,但短板仍然存在。其中一个短板是解析小于50-70kDa的没有明显的胞内或胞外结构域的单体膜蛋白,由于几乎没有胞外结构域特征,因此难以对去垢剂胶束或脂质纳米盘进行降噪处理,以这种方式收集到的数据难以产出高分辨率结构,比如解析没有上下游偶联蛋白的处于非活性状态的的GPCR结构。然而,大量的蛋白质属于这一类型,解析这一类型的的膜蛋白因此也成为了一个重要的研究领域。目前,有一些解决方案正处于研究阶段,且已经取得了一定程度的成功,如前文所述的CD20。随着利用增加融合蛋白、抗体片段、纳米抗体、纳米抗体衍生物或其他支架蛋白以增加靶点蛋白的分子量等方法的应用,预计冷冻电镜在膜蛋白结构测定方面会有更多进展。
计算赋能冷冻电镜
冷冻电镜单颗粒技术利用数百万个颗粒的可视化投影来重建静电势图,这通常涉及数十万亿字节的原始数据。因此,该方法从计算方法的快速发展中获益匪浅,这些计算方法同时满足了对更高的分辨率的需求并加深了对粒子动力学的理解。然而,与X射线晶体学相比,冷冻电镜在获取配体-靶点复合物的高可信度模型时仍然面临着一些难题。其中一个难题是冷冻电镜难以解析得到高于2.5Å的蛋白结构,而这通常是建模人员能够精确放置配体并解析出结合位点处水分子的最低分辨率。此外,冷冻电镜的结构建模流程与晶体学完全不同:在晶体学中,模型和密度图之间有一套严格而完善的统计测量方法,该方法能够提供和模型精度相关的关键信息。而在冷冻电镜方法中,基于密度图的建模是一个完全独立的过程,仅适用收集的电镜投影来进行密度图重构,然后基于密度图进行结构建模和实空间下的微调。该过程的独立性使得模型的精度被降低了。这一问题在最近已得到改善[38]。此外,两种方法之间还存在一些物理上的差异,如晶体学依赖电子密度图,而冷冻电镜依赖静电势图。这些差异加在一起,使得晶体学的模型验证工具无法应用于冷冻电镜模型。因此,我们可能需要为精确性开发一些新的指标。一种解决方案是使用强大的计算技术和精确的分子力场对大分子及其配体在冷冻电镜结构中的相互作用进行模拟。比如PHENIX软件包结合实空间和傅里叶空间微调和OPLS3e力场的分子动力学模型,从而生成生物分子和小分子的几何统计精修模型[39]。OPLS3e微调工具已经被整合进到我们(作者)的自研软件GemSpot,它将各种计算方法整合为一个工作流程,从而提高冷冻电镜密度图中配体位置的准确性(图3C)[40]。新的计算工具也推动冷冻电镜在基于片段的药物发现(Fragment-based drug discovery)中发挥作用,其中高溶解度的小片段化合物被浸泡在由多个不同结构的化合物组成的生物分子靶点中。解析复合体的结构可以解释配体与结合口袋之间关键位点的相互作用,然后可以将其组合成一个先导化合物。然而,这种方法要求配体密度质量高、分辨率高,才能正确区分配体的姿态和原子类型,目前对于冷冻电镜来说还是一个难题。最近,Saur等人在高度棘手的β-半乳糖苷酶和颇具治疗意义和挑战性的激酶PKM2的场景中成功地将冷冻电镜用于FBDD[41]。尽管他们为了将配体置放于密度图中,而不得不将干法和湿法实验结合,但他们成功地建立了一个与β-半乳糖苷酶结合的大约150kDa的精准片段模型。更令人印象深刻的是,他们能够从四种化合物的鸡尾酒中确定哪些片段与PKM2结合(图3, D)。因此,不断发展的计算方法为冷冻电镜密度图的构建提供了一个强大的平台,可以在高分辨率下对大分子复合物进行建模。
冷冻电镜的快速发展可及性与通量的提升
冷冻电镜是极为精密且昂贵的仪器,需要大量的费用和人力成本来搭建、维护与操作。这一特性在很大程度上限制了冷冻电镜的发展,并将冷冻电镜的机时资源集中在了那些受政府资金扶持的大型机构上。因此,在科研界中,冷冻电镜资源的获取门槛极高。然而,这一门槛正在被逐渐降低:许多国家级设施都启动了冷冻电镜人才培养计划,以降低冷冻电镜运维的人力成本。一些大型制药公司也开始进行内部投资,设立最先进的冷冻电镜设施。此外,冷冻电镜设施的可复制性远超晶体学极其昂贵的同步加速器和线性加速器,使得该技术更有发展前景。随着100kV电子束技术的发展[42],未来可能会出现性价比极高的冷冻电镜,增加其在药物发现领域中的应用场景。鉴于2018年FDA批准的药物中有49%来源于中小型公司,降低冷冻电镜的成本将使冷冻电镜技术得到更广泛的应用。最近对SARS-CoV-2相关蛋白的结构表征证明了冷冻电镜的无限潜力。在病毒爆发后的几个月内,科学家们利用冷冻电镜,以极快的速度解析了新冠病毒刺突蛋白的几种构象,以及它与人源血管紧张素转换酶或许多中和人源抗体片段的复合物的结构[35, 43-46]。最近获得FDA批准的用于治疗COVID-19的再利用药物瑞德西韦(Remdesivir)与SARS-CoV-2 RNA聚合酶结合的结构也已被冷冻电镜解析[47]。鉴于X射线晶体学一直是病毒RNA聚合酶结构测定的传统方法,对新冠病毒的冷冻电镜结构解析是一个颠覆性的创新,凸显了冷冻电镜的高时效性特点在快速反应研究中的应用。此外,冷冻电镜的分辨率仍在大幅提高,最近的一份报告指出,作为冷冻电镜的代表性复合物结构,去铁蛋白apoferritin的分辨率达到了1.25Å,该分辨率足以对单个原子进行精准定位,在某些情况下甚至可以解析氢原子和质子化态[48, 49]。毋庸置疑,在样品制备良好的情况下,冷冻电镜的不断改进将持续打破结构解析的分辨率记录。冷冻电镜在药物发现和开发方面的应用将进一步受益于该技术的全面自动化。在载网准备方面,一些自动化工具正在出现,以解决不可重复性和样品浪费的难题[50-52]。这些技术的改进不仅会提高自动化的程度和可及性,还可能解决冷冻电镜载网制备中的其他难题,如减少颗粒在空气及水中的暴露程度[53]。此外,机器学习方法和深度神经网络也是提高颗粒筛选速度和准确性的关键[54, 55]。这些自动化方法甚至有望在未来成为冷冻电镜的核心技术,从而推动冷冻电镜在药物发现领域的发展。主流硬件和软件的改进也有望提高冷冻电镜在SBDD领域的可及性。例如,更高效的检测设备能显著提高冷冻电镜的产能。在一个标准的数据收集过程中,老式的检测器相机可以每次收集1个影像,每小时产生50个影像,而较新的检测器可以每次收集9-16个影像,每小时可以产生超过200个影像,进而转化为每24小时收集的数百万颗粒投影数据。此外,虽然今天许多最高分辨率的结构是用300kV冷冻电镜获得的,但这些机器非常庞大,且前期和维护成本昂贵。在许多情况下,对于单颗粒分析中使用的薄样品,200kV的显微镜可能就足够了,甚至100kV的显微镜也可以用来获得分辨率高达3.4 Å的结构[42]。
分子动力学的新窗口
结合硬件和数据处理方面的改进,冷冻电镜的潜力将进一步被释放。当X射线晶体学受限于结晶条件而无法解析时,冷冻电镜的低样品需求大幅降低了数据收集的门槛,使我们得以看到样品的构象连续体或一系列不同的能量最低状态,为大分子动力学提供了新的窗口。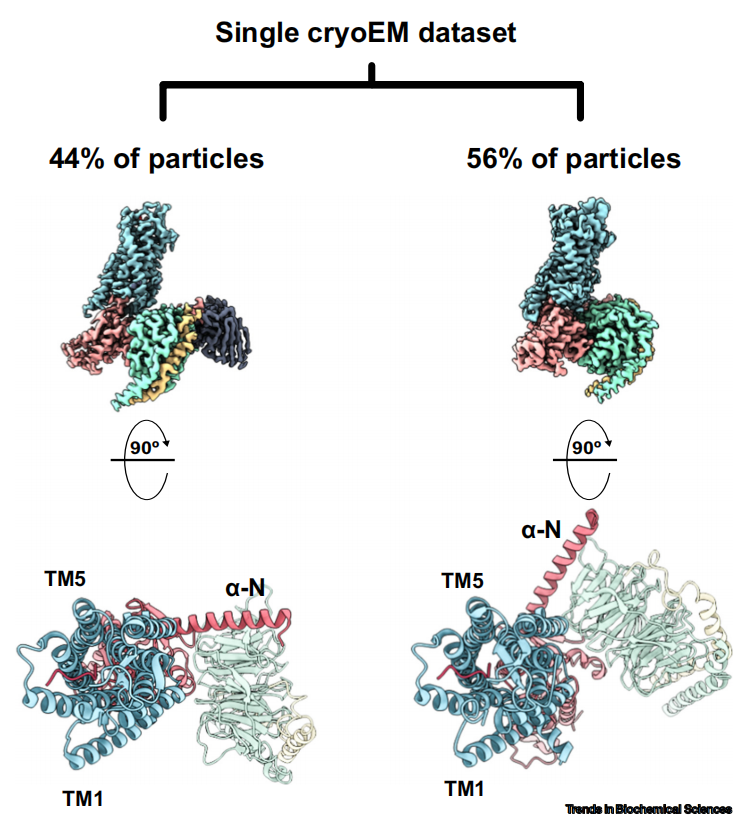
图4.单一的冷冻电镜数据集,投影的三维分类显示了两种不同的构象,代表了两种不同的G蛋白偶联受体-G蛋白相互作用的状态,代表了两种热力学上可比较的构象。在典型状态下(左边,PDB ID 6OS9),受体以典型的方式与G蛋白结合,其中核苷酸结合口袋为GTP结合做准备。在非经典状态下(右图,PDB ID 6OSA),G蛋白异源三聚体与经典状态相比旋转了45°,代表了沿G蛋白偶联途径的中间配体结合受体状态。缩写:α-N=G蛋白的N端α螺旋;cryo-EM=冷冻电镜;TM=跨膜螺旋。
一些计算工具,例如二维和三维分类以及子区域的重点细化,能够利用数据集内颗粒的异质性来模拟大分子活性成分的运动。在我们(作者)小组最近的一个例子中,对神经紧张素1受体的冷冻电镜单颗粒分析结果揭示了先前识别的G蛋白、激动剂结合状态和G蛋白偶联通路上的一个新的中间状态(图4)[57]。最近,我们(作者)还将AI深度学习网络应用于冷冻电镜数据集,揭示了26S蛋白酶体的构象动态,使解析出的结构细节达到了前所未有的原子级水平[58]。随着分辨率和分类工具的不断改进,我们将获得更精细的构象变化。有了以上这些技术,再加上分子动力学模拟和机器学习方法等计算技术,我们将得以对配体结合的复杂过程进行更精确的建模,从而揭示全新的、可成药的中间状态。
结语
尽管冷冻电镜已经在SBDD领域取得了飞跃性的进展,但它的潜力远不止于此。在三维分析重构及深度学习算法等领域,若能将计算工具与更大、更高质量的数据集结合并进行训练,我们将能够描述蛋白质甚至其配体的更小幅度、更高分辨率的动态运动。我们还期望冷冻电镜在时间维度上的结构解析方法将使人们对大分子复合物的结合和解离过程有更深了解,为靶向药物的研发提供更多思路和机会[59,60]。目前晶体学和大多数冷冻电镜结构所提供的只是能量最小值的瞬间结构,但对于开发新的药物作用模式而言,对机制和中间状态的理解至关重要,所以我们若能获取构象的动态信息,则对理性药物设计具有突破性意义。在综合了冷冻电镜的软硬件及方法的快速发展之后,我们可以得出结论:冷冻电镜有望为药物发现和人类健康做出巨大贡献。
以上文章转载于水木未来资讯
关于晶蛋生物
深圳晶蛋生物医药科技有限公司成立于2019年8月,致力于慢性肾病、抑郁症和恶性肿瘤等重大疾病的膜蛋白创新药物研发,立志成为全球极具创新力和影响力的企业,近日已完成近亿元Pre-A轮融资。
核心团队由哈佛大学、中科院上海药物所等多所顶尖科研院所和制药公司的博士组成,在膜蛋白抗原抗体、药物靶点结构解析、基于结构和人工智能的药物研发和药理学等领域的研发能力处于国际一流水平。
公司于2020年获得中国深圳创新创业大赛生物医药行业一等奖、2020年“科创中国”先导技术奖、第九届中国创新创业大赛全国优秀奖、科创中国科技创新创业大赛TOP10和粤港澳大湾区生物科技创新企业50强等奖项。
词表:
1. 激动剂
一种通过增加受体活性以产生生物反应的物质。
2. 拮抗剂(也称中性拮抗剂)
一种能阻断激动剂或反向激动剂的物质,在不存在激动剂或反向激动剂的情况下便没有活性
3. 生物制药
在生物活体中制造的药物,可能含有重组蛋白、糖类、基因疗法或核酸。
4. 电子密度图
电子密度与晶体中每一个晶胞位置的关系图,以二维或三维表示,由解析X射线衍射图案得出。
5. 静电势图(即库仑势图)
即样品中电荷分布的二维或三维表示。在电子显微镜中,静电势图由电子被与样品相互作用时产生的库仑力散射而产生。
6. 基于片段的药物发现FBDD
一种药物筛选方法,以评估小分子量的化学片段与期望靶点相结合的能力的方式,确认后续的药物化学实验方向。
7. 反向激动剂
一种通过减少受体的基础活性以产生生物反应的物质。
8. 脂质立方相结晶
基于脂立方相的蛋白结晶技术,采用脂立方相模拟生物膜环境,膜蛋白可以在脂质双分子层中相互接触,在合适的条件下形成晶体。
9. 脂质纳米盘
一个纽扣电池形状的盘状脂质双层,由两个环绕的两亲性螺旋蛋白(膜支架蛋白)稳定并使其可溶于水。
10. 冷冻电镜负染
一种将重金属盐染色剂嵌入并固定在生物标本上,并在室温下进行电子显微镜成像的技术方法。尽管只能在低分辨率(~2纳米)下观察标本的形状,但这种技术对于简单和快速评估样品质量是很有价值的。
11. 时间分辨的冷冻电镜方法
在动态构象转变的特定时间间隔内对生物样本进行速冻,并使用冷冻电镜技术进行可视化的方法。
参考资料:1. Byers, L.D. and Wolfenden, R. (1972) A potent reversible inhibitor of carboxypeptidase A. J. Biol. Chem. 247, 606–6082. Patlak, M. (2004) From viper’s venom to drug design: treating hypertension. FASEB J. 18, 4213. Baldwin, J.J. et al. (1989) Thienothiopyran-2-sulfonamides: novel topically active carbonic anhydrase inhibitors for the treatment of glaucoma. J. Med. Chem. 32, 2510–25134. Eder, J. et al. (2014) The discovery of first-in-class drugs: origins and evolution. Nat. Rev. Drug Discov. 13, 577–5875. Ostrem, J.M. et al. (2013) K-Ras(G12C) inhibitors allosterically control GTP affinity and effector interactions. Nature 503, 548–5516. Zhang, G. et al. (2015) Discovery of chemical inhibitors of human bromodomains. Chem. Rev. 115, 11625–11668 Trends in Biochemical Sciences7. Perera, T.P.S. et al. (2017) Discovery and pharmacological characterization of JNJ-42756493 (erdafitinib), a functionally selective small-molecule FGFR family inhibitor. Mol. Cancer Ther. 16, 1010–10208. Pan, S. et al. (2013) Discovery of BAF312 (Siponimod), a potent and selective S1P receptor modulator. ACS Med. Chem. Lett. 4, 333–3379. Ali, A. et al. (2010) Structure-based design, synthesis, and structure activity relationship studies of HIV-1 protease inhibitors incorporating phenyloxazolidinones. J. Med. Chem. 53, 7699–770810. Srivastava, A. et al. (2011) New target for inhibition of bacterial RNA polymerase: “switch region”. Curr. Opin. Microbiol. 14, 532–54311. Jia, Y. et al. (2016) Overcoming EGFR(T790M) and EGFR (C797S) resistance with mutant-selective allosteric inhibitors. Nature 534, 129–13212. Danev, R. et al. (2019) Cryo-electron microscopy methodology: current aspects and future directions. Trends Biochem. Sci. 44, 837–84813. Renaud, J.-P. et al. (2018) Cryo-EM in drug discovery: achievements, limitations and prospects. Nat. Rev. Drug Discov. 17, 471–49214. McGrath, N.A. et al. (2010) A graphical journey of innovative organic architectures that have improved our lives. J. Chem. Educ. 87, 1348–134915. Shalev-Benami, M. et al. (2017) Atomic resolution snapshot of Leishmania ribosome inhibition by the aminoglycoside paromomycin. Nat. Commun. 8, 158916. Qu, Q. et al. (2018) Structure and conformational dynamics of a COMPASS histone H3K4 methyltransferase complex. Cell 174, 1117–1126 e1217. Wang, H. et al. (2020) Structure of the transcription coactivator SAGA. Nature 577, 717–72018. Bonekamp, N.A. et al. (2020) Small-molecule inhibitors of human mitochondrial DNA transcription. Nature 588, 712–71619. Kruse, A.C. et al. (2013) Activation and allosteric modulation of a muscarinic acetylcholine receptor. Nature 504, 101–10620. Waltenspühl, Y. et al. (2020) Crystal structure of the human oxytocin receptor. Sci. Adv. 6, eabb541921. Thonghin, N. et al. (2018) Cryo-electron microscopy of membrane proteins. Methods 147, 176–18622. Gao, Y. et al. (2016) TRPV1 structures in nanodiscs reveal mechanisms of ligand and lipid action. Nature 534, 347–35123. Dang, S. et al. (2019) Structural insight into TRPV5 channel function and modulation. Proc. Natl. Acad. Sci. U. S. A. 116, 8869–887824. Masiulis, S. et al. (2019) GABAA receptor signalling mechanisms revealed by structural pharmacology. Nature 565, 454–45925. Alam, A. et al. (2019) Structural insight into substrate and inhibitor discrimination by human P-glycoprotein. Science 363, 753–75626. Staus, D.P. et al. (2020) Structure of the M2 muscarinic receptor-β-arrestin complex in a lipid nanodisc. Nature 579, 297–30227. Denisov, I.G. and Sligar, S.G. (2017) Nanodiscs in membrane biochemistry and biophysics. Chem. Rev. 117, 4669–471328. Efremov, R.G. et al. (2017) Lipid nanodiscs as a tool for high resolution structure determination of membrane proteins by single-particle cryo-EM. Methods Enzymol. 594, 1–3029. Yao, X. et al. (2020) Cryo-EM analysis of a membrane protein embedded in the liposome. Proc. Natl. Acad. Sci. U. S. A. 117, 18497–1850330. Pang, S.S. et al. (2019) The cryo-EM structure of the acid activatable pore-forming immune effector Macrophage-expressed gene 1. Nat. Commun. 10, 428831. Saeedi, P. et al. (2019) Global and regional diabetes prevalence estimates for 2019 and projections for 2030 and 2045: results from the International Diabetes Federation Diabetes Atlas, 9th edition. Diabetes Res. Clin. Pract. 157, 10784332. Scapin, G. et al. (2018) Structure of the insulin receptor-insulin complex by single-particle cryo-EM analysis. Nature 556, 122–12533. Uchikawa, E. et al. (2019) Activation mechanism of the insulin receptor revealed by cryo-EM structure of the fully liganded receptor-ligand complex. eLife 8, e4863034. Hao, Y. et al. (2019) Cryo-EM Structure of HER2-trastuzumabpertuzumab complex. PLoS One 14, e021609535. Pinto, D. et al. (2020) Cross-neutralization of SARS-CoV-2 by a human monoclonal SARS-CoV antibody. Nature 583, 290–29536. Rougé, L. et al. (2020) Structure of CD20 in complex with the therapeutic monoclonal antibody rituximab. Science 367, 1224–123037. Kumar, A. et al. (2020) Binding mechanisms of therapeutic antibodies to human CD20. Science 369, 793–79938. Yamashita, K. et al. (2021) Cryo-EM single particle structure refinement and map calculation using Servalcat. bioRxiv Published online May 4, 2021. https://doi.org/10.1101/2021.05.04.44249339. van Zundert, G.C.P. et al. (2020) Macromolecular refinement of X-ray and cryo-electron microscopy structures with Phenix / OPLS3e for improved structure and ligand quality. bioRxiv Published online August 21, 2021. https://doi.org/10.1101/2020.07.10.19809340. Robertson, M.J. et al. (2020) GemSpot: a pipeline for robust modeling of ligands into cryo-EM maps. Structure 28, 707–716 e341. Saur, M. et al. (2020) Fragment-based drug discovery using cryo-EM. Drug Discov. Today 25, 485–49042. Naydenova, K. et al. (2019) CryoEM at 100 keV: a demonstration and prospects. IUCrJ 6, 1086–109843. Chi, X. et al. (2020) A neutralizing human antibody binds to the N-terminal domain of the Spike protein of SARS-CoV-2. Science 369, 650–65544. Liu, L. et al. (2020) Potent neutralizing antibodies against multiple epitopes on SARS-CoV-2 spike. Nature 584, 450–45645. Walls, A.C. et al. (2020) Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein. Cell 181, 281–292 e646. Yan, R. et al. (2020) Structural basis for the recognition of SARSCoV-2 by full-length human ACE2. Science 367, 1444–144847. Kokic, G. et al. (2021) Mechanism of SARS-CoV-2 polymerase stalling by remdesivir. Nat. Commun. 12, 27948. Nakane, T. et al. (2020) Single-particle cryo-EM at atomic resolution. Nature 587, 152–15649. Yip, K.M. et al. (2020) Atomic-resolution protein structure determination by cryo-EM. Nature 587, 157–16150. Ravelli, R.B.G. et al. (2020) Cryo-EM structures from sub-nl volumes using pin-printing and jet vitrification. Nat. Commun. 11, 256351. Klebl, D.P. et al. (2020) Need for speed: examining protein behavior during cryoEM grid preparation at different timescales. Structure 28, 1238–1248 e452. Rubinstein, J.L. et al. (2019) Shake-it-off: a simple ultrasonic cryo-EM specimen-preparation device. Acta Crystallogr. 75, 1063–107053. Noble, A.J. et al. (2018) Reducing effects of particle adsorption to the air-water interface in cryo-EM. Nat. Methods 15, 793–79554. Al-Azzawi, A. et al. (2020) DeepCryoPicker: fully automated deep neural network for single protein particle picking in cryo-EM. BMC Bioinforma. 21, 50955. Wagner, T. et al. (2019) SPHIRE-crYOLO is a fast and accurate fully automated particle picker for cryo-EM. Commun. Biol. 2, 21856. Nakane, T. et al. (2018) Characterisation of molecular motions in cryo-EM single-particle data by multi-body refinement in RELION. eLife 7, e3686157. Kato, H.E. et al. (2019) Conformational transitions of a neurotensin receptor 1-Gi1 complex. Nature 572, 80–8558. Wu, Z. et al. (2020) Deep manifold learning reveals hidden dynamics of proteasome autoregulation. bioRxiv Published online December 22, 2020. https://doi.org/10.1101/2020.12.22.42393259. Mäeots, M.-E. et al. (2020) Modular microflfluidics enables kinetic insight from time-resolved cryo-EM. Nat. Commun. 11, 346560. Dandey, V.P. et al. (2020) Time-resolved cryo-EM using Spotiton. Nat. Methods 17, 897–90061. Berman, H.M. (2000) The Protein Data Bank. Nucleic Acids Res. 28, 235–24262. Jo, S. et al. (2008) CHARMM-GUI: A web-based graphical user interface for CHARMM. J. Comput. Chem. 29, 1859–186563. Pettersen, E.F. et al. (2021) UCSF CHIMERAX : structure visualization for researchers, educators, and developers. Protein Sci. 30, 70–82